Die Bestimmung des Serotyps von Actinobacillus pleuropneumoniae (App) ist wichtig für die Bekämpfung der Krankheit, da verschiedene Serotypen (je nach geografischer Region) unterschiedliche Virulenzpotenziale haben und die Informationen dazu dienen können, den geeignetsten Impfstoff auszuwählen (Gottschalk, 2015). Beruhend auf ihrer Hülle kennt man derzeit 18 Serotypen von App (Bossé et al., 2018a), wobei Serotyp 1, 5, 9 und 11 am virulentesten sind. Aufgrund des Bedarfs an NAD (Nicotinamidadenindinukleotid) kann man Biotyp 1 und die NAD-unabhängigen Biotyp-2-Isolate unterscheiden.
Klassischerweise wurden serologische Tests einschließlich der Agglutination, Koagglutination, Immundiffusion, der indirekten Hämagglutination und der Ringpräzipitation verwendet, um den Serotyp zu bestimmen. Hierzu sind jedoch hochtitrige spezifische Antiseren erforderlich und Kreuzreaktionen zwischen Serotypen (z. B. 3/6/8 und 1/9/11) sind problematisch. Wir fanden heraus, dass die meisten UK- und Irland-Isolate, die serologisch dem Serotyp 3 zugeordnet wurden, tatsächlich zum Serotyp 8 gehörten (O'Neill et al., 2010). Aufgrund solcher Probleme haben wir mithilfe der Polymerase-Kettenreaktion (PCR) molekularbasierte Tests entwickelt, um in den Genen zur Biosynthese der Bakterienhülle serotypspezifische DNA-Sequenzen zu amplifizieren (Bossé et al., 2014). Die Ausrichtung auf diese serotypspezifischen Sequenzen ermöglichte die Entwicklung eines Serotyp-16-spezifischen Tests (Bossé et al., 2017) und führte kürzlich zur Entdeckung der Serotypen 17 und 18 (Bossé et al., 2018a). Aufgrund der Entdeckung der Serotypen 16-18 entschieden wir uns, einen PCR-basierten Test zu entwickeln, der alle bekannten App-Serotypen zuordnen kann. Die Serotypen 9 und 11 können jedoch weder durch PCR unterschieden werden, da ihre kapselförmigen Loci nahezu identisch sind (Bossé et al., 2018b), noch kann man sie durch serologische Tests voneinander trennen (Gottschalk, 2015). Der PCR-basierte Test musste das Isolat als App bestätigen und den spezifischen Serotyp bestimmen. Zur Bestätigung von App verwendeten wir Primer, die eine 418bp-Region des App-spezifischen apxIV-Gens amplifizierten (Schaller et al., 1999). Aufgrund der großen Anzahl an Serotypen (n=18) mussten wir jedoch zwei Multiplex-PCR-Tests (mPCR) entwickeln, die jeweils in der Lage waren, viele Serotypen zu erkennen. Der mPCR1-Test erkennt die Serotypen 1-12 und 15 (Abb. 1A) und der mPCR2-Test die Serotypen 13-14 und 16-18 (Abb. 1B). Isolate, die nur eine apxIV-Bande im mPCR1-Test amplifizieren, werden im mPCR2-Test weiter getestet.


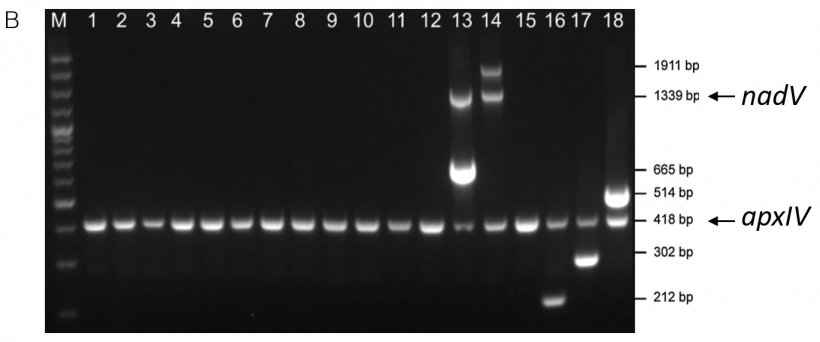
Der mPCR2-Test bestätigt den Biotyp auch durch Primer, die ein 1339bp-Fragment des nadV-Gens amplifizieren (das für die Unabhängigkeit von NAD verantwortlich ist). Das 1339bp-nadV-Amplikon wird nur in den Referenzstämmen des Biotyps 2 (Serotypen 13-14) nachgewiesen. Es sei darauf hingewiesen, dass andere Serotypen (z. B. Serotyp 2, 4, 7 und 17) als Biotyp 2 und einige nordamerikanische Serotyp 13-Isolate als Biotyp 1 beschrieben wurden (Gottschalk, 2015).
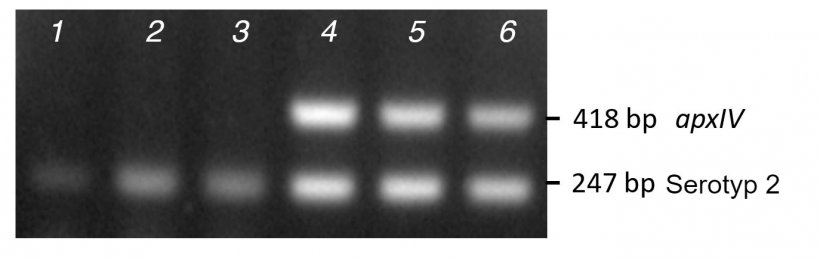
Die DNA-Vorlage für die PCRs kann gereinigte DNA (aus kultivierten Bakterien oder Gewebeproben) sein, die mit kommerziellen Kits, gekochten bakteriellen Ganzzelllysaten oder Kolonien aus einer Kulturschale gewonnen wird. Gereinigte DNA liefert die besten Ergebnisse, während Kolonie-PCRs Teil- bzw. falsch negative Ergebnisse liefern können, wenn Kolonien sehr klebrig und schwer zu lysieren sind, wie bei drei klinischen Serotyp-2-Isolaten zu sehen ist, die zwar nur die serotypspezifische Bande aus der Kolonie-PCR amplifizierten, aber mit gereinigter DNA sowohl diese als auch die apxIV-spezifische Bande amplifizierten (Abb. 2). Selbst wenn gereinigte DNA verwendet wird, schaffen es Isolate gelegentlich nicht, apxIV zu amplifizieren (Bossé et al., 2014). In diesem Fall können alternative apxIV-Primer (oAPXIV-TSP1/2) verwendet werden (Tegetmeyer et al., 2008), um App zu bestätigen. Der Einsatz von mPCR1- und mPCR2-Tests ist sinnvoll, um neue Serotypen zu identifizieren, wie wir es im Falle von Serotyp 17 und 18 getan haben (Bossé et al., 2018a). App-Isolate, die eine apxIV-Bande erzeugen, aber keine serotypspezifischen Amplikone, könnten ein neuer Serotyp sein, obwohl das Fehlen einer serotypspezifischen Bande möglicherweise auf eine mangelnde Übereinstimmung des Primers in unterschiedlichen Isolaten oder auf ein bestehendes Insertionselement zurückzuführen ist, das den Locus der Hülle verändert. Die vollständige Genomsequenzierung des Isolats wird bestätigen, welche dieser Möglichkeiten zutrifft.
Zusammenfassend ist die Kontrolle der App-Serotypen in einem Betrieb oder einem Land wichtig, um Krankheiten zu bekämpfen. Unsere serotypisierenden PCRs (mPCR1 und mPCR2) sind nützliche Werkzeuge, um virulente Serotypen zu identifizieren, die richtigen (handelsüblichen oder eigenen) Impfstoffe zu finden und zu verhindern, dass Schweine mit potenziell virulenten App-Isolaten in naive Betriebe eingeführt werden. Darüber hinaus hat unser Ansatz das Potenzial, neue App-Serotypen zu identifizieren und eine verbesserte Diagnose zu ermöglichen.
Danksagungen
Die App-Forschung im Labor des Autors wird vom Biotechnology and Biological Sciences Research Council des Vereinigten Königreichs unterstützt.




