Schweineinfluenzaviren (SIV) gehören zur Gattung Influenzavirus Typ A, Familie Orthomyxoviridae. Diese Viren werden weiterhin auf Grundlage der Oberflächenglykoproteine Hämagglutinin (HA) und Neuraminidase (NA) klassifiziert, von denen 17 bzw. 10 verschiedene Typen beschrieben sind. Die Kombination eines HA-Typs mit einem NA-Typ definiert den Influenza-Subtyp. Die wichtigsten Subtypen bei Schweinen sind demgemäß H1N1, H1N2 und H3N2.
Gemäß verschiedenen Studien, die in Europa durchgeführt wurden, ist die Seroprävalenz der drei Subtypen (H1N1, H1N2 und H3N2) in denjenigen Ländern sehr hoch, in denen intensive Schweinehaltung betrieben wird. In Belgien, Dänemark und Spanien liegt die Seroprävalenz der drei Subtypen z. B. über 30%. In Spanien wurden 94% der Betriebe auf mindestens einen der Subtypen positiv getestet. In Frankreich und Großbritannien konnte eine hohe Prävalenz von H1N1 oder H1N2 nachgewiesen werden. Allerdings wurde der Subtyp H3N2 in letzter Zeit nicht gefunden. In anderen Ländern wie der Tschechischen Republik, Irland und Polen scheint die Prävalenz des SIV niedriger zu sein.

Im selben Bestand kann mehr als ein SIV-Subtyp direkt (PCR + Sequenzierung) oder indirekt (durch Hämagglutinationshemmung) nachgewiesen werden, wie man unter anderem in Betrieben in den Niederlanden, Frankreich und Spanien festgestellt hat. Einzeln betrachtet kann der Anteil von Tieren mit Antikörpern gegen mehr als einen Subtyp sowohl bei Sauen als auch bei Mastschweinen hoch sein. Dies deutet darauf hin, dass eine SIV-Infektion aktiv ist und häufig vorkommt. Dessen ungeachtet ist das Virus nicht immer mit dem Ausbruch von Atemwegserkrankungen verbunden. Tatsächlich gibt es eine hohe Zahl an positiv getesteten Betrieben ohne offensichtliche klinische Symptome, die durch das Virus verursacht wären. Kurz gesagt legen diese Befunde nahe, dass SIV in hohem Maße in Schweinemastbetrieben vorhanden ist und zu einer enzootischen und offensichtlich subklinischen Infektion führt.
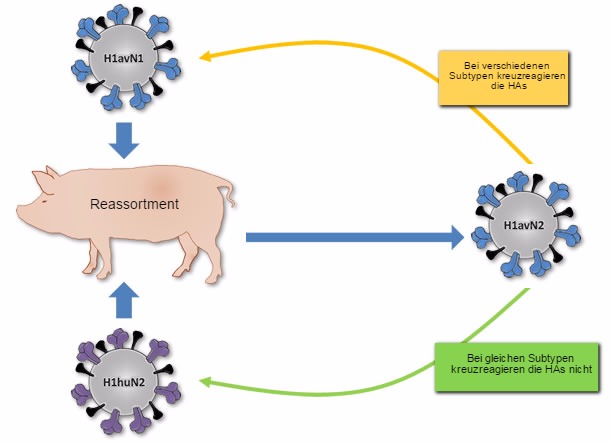
Abbildung 1: Herkunft des in Dänemark und Frankreich beschriebenen H1avN2-Virus. Dieses Virus entstand durch Reassortment des H1avN1-Virus (HA-Donor) und des H1huN2-Virus (NA-Donor). H1avN2 und H1avN1 gehören deshalb zu verschiedenen Subtypen, aber ihre HAs kreuzreagieren. H1avN2 und H1huN2 gehören zum selben Subtyp, aber ihre HAs kreuzreagieren nicht.
Die Klassifikation der Subtypen ist zwar sinnvoll um die Epidemiologie und die Diversität des SIV zu vereinfachen und zu verstehen, die SIV-Diversität ist allerdings weitaus komplexer. Die genetische Vielfalt des europäischen SIV wurde von Dr. Simon weitestgehend zusammengefasst . Kurz gesagt können wir in Europa in Abhängigkeit vom HA-Typ mindestens vier Linien unterscheiden: 1) Viren, die mit dem avian-like H1N1-Typ (H1avN1) verwandt sind, 2) Viren, die mit dem reassortierten human-like H1N2 (H1huN2) verwandt sind, 3) Viren, die mit dem pandemischen H1N1 (H1N1pdm) verwandt sind, und schließlich 4) die H3N2-Viren.
Wir dürfen nicht vergessen, dass SIV ebenso wie andere Viren, die zur Gattung des Influenzavirus A gehören, aus einem Genom besteht, das in 8 RNA-Stücke segmentiert ist, wodurch sich das Genom mithilfe von zwei Mechanismen verändern kann: 1) Drift: Mutationen ereignen sich an besonderen Stellen des HA (überwiegend) oder der NA. Dadurch können virale Varianten entstehen, die sich der Immunität entziehen können, die zuvor gegen SIV-Vorgänger entwickelt wurde (Hauptursache der saisonalen Grippe beim Menschen). 2) Reassortment: Dies geschieht, wenn zwei oder mehr Fragmente von RNA-Viren ausgetauscht werden (z. B. die Entstehung des H1huN2-Stamms im Jahr 1994 als Folge des Reassortments zwischen einem humanen H1N1- und einem porzinen H3N2-Virus).
Obwohl die drei verschiedenen H1-Viren (H1avN1, H1huN2 und H1N1pdm) zu einem früheren Zeitpunkt einen gemeinsamen Vorgänger hatten, zeigen sie keine Kreuzreaktion auf einem antigenischen Niveau bzw. tun dies kaum. Der Grund dafür liegt wahrscheinlich darin, dass sich die drei H1-Varianten durch Drift zu drei verschiedenen Varianten entwickelt haben, bevor das Schweinefleisch nach Europa kam, nämlich das H1avN1 bei Vögeln, das H1huN2 beim Menschen und das H1N1pdm bei Schweinen in Nordamerika. Zudem kann Reassortment neue Influenzavirus-Stämme hervorbringen, die, obwohl sie zu einem bestimmten Subtyp gehören, antigenisch mit Viren kreuzreagieren können, die zu anderen Subtypen gehören. Ein gutes Beispiel hierfür ist der H1avN2-Stamm, der in Dänemark und Frankreich beschrieben wurde. Dieser Stamm enthält ein HA, das mit H1avN1-Stämmen verwandt ist, und kreuzreagiert antigenisch folglich nicht mit dem H1huN2-Stamm, aber mit den H1avN1-Stämmen (s. Abb. 1).
Die genetische and antigenische Diversität des Influenzavirus muss als etwas Dynamisches verstanden werden, das sich ständig verändert. Diese Auffassung wurde in Studien nachgewiesen, die in jüngster Zeit in Deutschland, Dänemark, Spanien und Italien durchgeführt wurden. In diesen Studien wurden neue SIV-Stämme isoliert und beschrieben und es wurde gezeigt, dass sie das Produkt des Reassortments zwischen SIV-Stämmen und humanen Viren der saisonalen Grippe sind. Aus den genannten Gründen ist es deshalb entscheidend, die aktive Überwachung des SIV beizubehalten und noch weiter zu fördern, um unsere Kenntnisse über die bei europäischen Schweinen vorkommenden SIV-Stämme und deren jeweilige Prävalenz und Auswirkungen auf die Schweinemast zu verbessern.


