E. coli (EC) – Kultureller Nachweis und Serotypisierung
Das gramnegative Stäbchenbakterium tritt in einer Vielzahl an Varianten auf. Es werden kommensale, also natürlich bei allen Säugetieren in der Darmflora vorkommende E. coli, aber auch intestinal pathogene (InPEC) und extraintestinal pathogene E. coli (ExPEC) unterschieden. Die Unterscheidung dieser Varianten ist allerdings seit je her ein diagnostisches Problem, da die Differenzierung von Pathotypen oder Pathovaren mit einfachen phänotypischen Methoden nicht möglich ist.

Der Nachweis von E. coli gelingt relativ unproblematisch aus geeignetem Probenmaterial (Dünndarm, Kot) von akut erkrankten Schweinen durch die kulturelle Anzüchtung auf Blutagar (s. Abbildung 1) und auf Differenzialnährböden (z.B. Gassner- oder MacConkey-Agar). Zur Differenzierung von E. coli-Isolaten hat sich in der Vergangenheit die Typisierung durch Bestimmung von Serotypen oder Serovaren bewährt, da bestimmte Serotypen häufig mit bestimmten Erkrankungen assoziiert sind (s. Tabelle 1). In der Tiermedizin werden daher gewöhnlich die O- Antigene (Bestandteile des LPS [auch Endotoxin] der bakteriellen Zellwand) und die F- oder Fimbrien-Antigene (Adhäsine, die die wirtsspezifische Anheftung an Dünndarmepithelzellen und die Besiedlung vermitteln) mittels Objektträgerschnellagglutination unter Verwendung von poly- oder monovalenten Seren bestimmt. Die Methode ist schnell aber auch fehleranfällig, da es durch Kreuzreaktionen, Maskierungen der Zielantigene oder fehlender Fimbrienausbildung in vitro zu Fehlklassifizierungen kommen kann. Für eine vollständige Serotyp-Bestimmung wird dagegen seit Jahrzehnten und immer noch die O:K:H-Seroformel verwendet, d.h. es werden neben den O-Antigenen auch die K-Antigene (Kapselpolysaccharid-Antigene) und die H-Antigene (Geißelprotein-Antigene) bestimmt.
Tabelle 1: Intestinal pathogene E. coli beim Schwein
| Pathotyp | E. coli bedingte Erkrankungen | Häufig assoziierte O-Antigen-Serotypen | Häufig nachgewiesene Fimbrien-Serotypen |
| Intestinal pathogene E. coli (InPEC) | E. coli-Diarrhoe der Saugferkel | O8, O108, O138, O139, O141, O147, O149 und O157 | F4, F5, F6 und F41 |
| E. coli-Diarrhoe der Absetzferkel | O8, O108, O138, O139, O141, O147, O149 und O157 | F4, F18ac | |
| Ödemkrankheit der Absetzferkel | O138, O139 und O141 (O147, O157 und routinemäßig nicht serotypisierbare) | F18ab |
E. coli (EC) – Pathotypen, Virotyping oder Virotypisierung
Aufgrund der Weiterentwicklung molekularbiologischer Methoden und der Fortschritte in der molekularbiologischen Analyse von E. coli sind inzwischen viele Gene bekannt, die für Virulenzfaktoren kodieren (s. Tabelle 2). Dabei handelt es sich zum einen um klassische Virulenzfaktoren mit schädigender Wirkung (Toxine) und zum anderen um Faktoren, die die Besiedlung und das Überleben von E. coli im Wirt direkt oder indirekt vermitteln. Anhand des Nachweises dieser Gene, z.B. mittels PCR- oder Array-Verfahren, ist es inzwischen möglich, das Virulenzpotenzial von E. coli-Isolaten zu identifizieren und verschiedene Pathotypen zu differenzieren (s. Tabelle 3). Man nennt diese Verfahren zur Bestimmung von Virulenzmarkern, also Zielgenen für Virulenzfaktoren, auch Englisch Virotyping oder Virotypisierung.
Tabelle 2: Beispiele für bei E. coli nachweisbare Zielgene und Virulenzfaktoren (Fimbrien, Adhäsine und Toxine), für die sie kodieren
| Zielgene | Virulenzfaktoren |
| faeG (F4) | F4-Fimbrien |
| fanC (F5) | F5-Fimbrien |
| fasA (F6) | F6-Fimbrien |
| fedA (F18) | F18-Fimbrien |
| fim41A (F41) | F18-Fimbrien |
| fimH (F1) | Typ-1-Fimbrien |
| fimA (F1) | Typ-1-Fimbrien |
| papC | P-Fimbrien |
| aidA (AIDA) | AIDA-I-Autotransporter Adhäsin |
| paa | porcine adherence factor |
| eaeA (intimin) | intimin |
| eltB (LTI) | hitzelabiles Enterotoxin |
| estA (STI) | hitzestabiles Enterotoxin |
| est B (STII) | hitzestabiles Enterotoxin |
| estA (EAST) | hitzestabiles Enterotoxin |
| stx2e | Shigatoxin Variante 2e |
| cdtB | cytolethal distending toxin |
| cnf1 | zytotoxisch nekrotisierender Faktor Typ 1 |
| iucD | Aerobactin |
| escV | Typ-III-Sekretionssystem |
| pic | Serinprotease Autotransporter |
Tabelle 3: Zielgene zur Identifizierung von E. coli-Pathotypen beim Schwein
| Pathotypen | Fimbrien und Adhäsine (Zielgene) |
Toxine (Zielgene) | Symptome | |
Intestinal pathogene E. coli (InPEC) |
Enteropathogene E. coli (EPEC) | fimA/fimH paa intimin |
escV pic cdtB |
Diarrhoe durch Malabsorption |
| Enterotoxische E. coli (ETEC) | F4, F5, F6, F18, F41 fimA/fimH AIDA paa |
STI, STII, EAST LTI Stx2e Hämolysin |
Diarrhoe durch Hypersekretion |
|
| Ödemkrankheits (edema disease)-E. coli (EDEC) | F18 AIDA |
Stx2e Hämolysin |
der Ödemkrankheit | |
| Shigatoxin- bildende E. coli (STEC) | fimA, fimH intimin |
Stx2e EAST escV pic, cdtB Hämolysin |
Katarrhalisch, hämorrhagische Diarrhoe |
|
| Extraintestinal pathogene E. coli (ExPEC) |
Septikämische E. coli (SEPEC) | papC | iucD | Hämorrhagische Septikämie |
| Uropathogene E. coli (UPEC) | papC fimA, fimH | cnf1, iucD Hämolysin |
Coliforme Mastitis der Sau |
|
Dabei tragen pathogene E. coli-Isolate grundsätzlich eine Kombination aus Fimbrien und Adhäsinen sowie Toxinen. Es können mehrere Toxine und Adhäsine zugleich und in unterschiedlichen Kombinationen vorliegen. Durch den Nachweis der verschiedenen Virulenzmarker bzw. den Nachweis Ihrer Gene erhält man Hinweise auf den Pathotyp kulturell nachgewiesener E. coli-Isolate (s. Tabelle 3). Hervorzuheben ist, dass nicht nur hämolysierende E. coli pathogen sind (s. Abbildung 1), sondern gerade E. coli ohne Hämolyse häufig im ursächlichen Zusammenhang mit klinischen Erkrankungen nachzuweisen sind. Ferner besitzen hämolysierende E. coli nicht generell relevante Fimbrien- und Toxin-Gene. Daher sollten möglichst alle Varianten der kulturell nachgewiesenen E. coli-Isolate virotypisiert werden, um sicher zu stellen, dass auch der ursächliche Erreger identifiziert werden kann. Informationen zu den Fimbrien-Typen und Toxinen nachgewiesener pathogener E. coli sind auch hilfreich bei der Auswahl geeigneter Impfstoffe mit entsprechenden Impfantigenen.
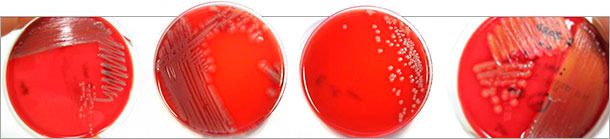
Abbildung 1: Verschiedene Koloniemorphologien auf Blutagar (Columbia-Agar) von nach Virotypisierung mittels Multiplex-PCR identifizierten pathogenen E. coli-Isolaten. Nicht nur hämolysierende E. coli (rechts) sind pathogen.

Bei der Einschätzung diagnostischer Befunde ist allerdings zu beachten, dass E. coli-Pathotypen natürlich auch von gesunden Wirten isoliert werden können (latente Infektion), insbesondere aus dem Habitat Darm. Eine Bewertung diagnostischer Befunde kann daher nur unter Berücksichtigung sowohl der Klinik des jeweiligen Tieres als auch der Konzentration der isolierten Keime im Zusammenhang mit dem nachgewiesenen Pathotyp erfolgen.
Schwierig wird die Interpretation der Virotypisierung, wenn die Virulenzmarker am Probenmaterial, z.B. Kot, direkt nachgewiesen werden und keine Typisierung der einzelnen Isolate erfolgt. Das ist zwar technisch möglich und wird auch angeboten; wenn allerdings verschiedene E. coli-Pathotypen in der Probe enthalten sind, erhält man auch eine Mischung aller nachweisbaren Faktoren, die möglicherweise falsche Pathotypen identifizieren lassen und deren Kombination von Virulenzmarkern so real gar nicht vorliegt. Ferner kann es auch zum Nachweis von Virulenzmarkern von anderen in der Kotprobe vorkommenden Darmbakterien kommen, da es ähnliche Gene auch bei anderen Darmbakterien gibt. Dann werden vermeintliche Virulenzmarker für E. coli nachgewiesen, die aber gar keine Virulenzfaktoren von den in der Probe befindlichen E. coli sind. Virotypisierungsergebnisse an Proben sind daher mit besonderer Vorsicht zu bewerten.
Im Zusammenhang mit der klinischen und häufig auch notwendigen pathologischen Untersuchung, die hier bereits ausführlich an anderer Stelle beschrieben wurden, sowie den entsprechenden Differenzialdiagnosen trägt die Virotypisierung von E. coli-Isolaten allerdings zur deutlichen Verbesserung bei der Diagnostik von E. coli-Infektionen beim Schwein bei.


